Tabla de contenido:
- Paso 1: uso de Brain Box
- Paso 2: Introducción: análisis de secciones transversales
- Paso 3: Configurar una función: LevelCurveTracings.m
- Paso 4: Desarrollar el algoritmo de límites: encontrar límites
- Paso 5: desarrollo del algoritmo enlazado: generación de una matriz enlazada externa
- Paso 6: Desarrollar el algoritmo delimitado: trabajar con el punto central
- Paso 7: Desarrollo del algoritmo ligado: cuando un centroide no está centrado
- Paso 8: Desarrollo del algoritmo ligado: interferencia de agujeros
- Paso 9: Desarrollo del algoritmo delimitado: localización de los agujeros, finalización de los límites del cerebro y los límites del agujero
- Paso 10: Registro de datos: Función PatientFiles.m
- Paso 11: registrar datos en archivos
- Paso 12: Registro de datos: visualización de un gráfico del volumen cerebral a lo largo del tiempo
- Paso 13: Cerrar brechas en subparcelas: Subplotclose.m
- Paso 14: El código principal: borrar todo y solicitar entradas
- Paso 15: El código principal: procesamiento por lotes de las imágenes
- Paso 16: El código principal: relleno
- Paso 17: El código principal: Determinación de límites
- Paso 18: El código principal: determinar la dimensión Z adecuada
- Paso 19: El código principal: determinación de las coordenadas X e Y
- Paso 20: El código principal: trazar una estructura tridimensional, encontrar volumen y registrar datos
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-31 10:18.
- Última modificación 2025-01-23 14:40.
El avance hacia la frontera de una vida humana más larga ha provocado el surgimiento de enfermedades nunca vistas por civilizaciones anteriores a la nuestra. Entre estos, el Alzheimer afectó a aproximadamente 5,3 millones de estadounidenses ancianos vivos en 2017, o aproximadamente a 1 de cada 10 estadounidenses ancianos (https://www.alz.org/facts/) y a muchos otros con demencia. Para ayudar en la lucha por comprender lo que aflige a nuestros mayores, este código equipará a los futuros investigadores y a los curiosos ansiosos con la capacidad de rastrear el volumen cerebral a lo largo del tiempo.
Paso 1: uso de Brain Box
Para usar la caja del cerebro, solo se necesita lo siguiente:
- Imágenes por resonancia magnética de un cerebro y el nombre y formato de dichos archivos (todos deben tener aproximadamente las mismas dimensiones)
- Duración de un escaneo
- Distancia entre cada capa (resonancia magnética)
- Nombre del paciente (al ingresar, no incluya espacios y escriba en mayúscula el nombre y apellido, como tal: Nombre y apellido)
Y a partir de esto, uno tiene la capacidad de realizar un seguimiento de las tendencias individuales en el volumen cerebral a lo largo del tiempo. Por lo tanto, este software puede rastrear las cifras de las tendencias de la enfermedad de Alzheimer. La longitud que usamos en el ensayo fue de 180 mm para la longitud de una exploración y de 5 mm para la distancia entre exploraciones de resonancia magnética, según las cifras promedio.
Sin embargo, la aplicación de la caja del cerebro no tiene por qué limitarse a esta única tarea. Si las secciones transversales de un sólido dado se fotografían, como un tumor en sí mismo, las tendencias en los cambios de volumen para estos también se pueden rastrear en el software.
Paso 2: Introducción: análisis de secciones transversales
En estructuras tridimensionales, los planos bidimensionales que los componen se denominarían secciones transversales. Imagina que una pila de papeles forma un prisma rectangular, luego cada hoja de papel sería una sección transversal del papel. Al imaginar el cerebro, aplicamos el mismo curso de pensamiento. La resonancia magnética (imagen por resonancia magnética) (ver información sobre la resonancia magnética) captura las secciones transversales del cerebro y, al usar los límites definidos en cada "capa" del cerebro proporcionada, podemos construir una estructura para modelar y encontrar el volumen del cerebro.. Sin embargo, primero debemos construir una función para proporcionar información sobre dichos límites.
Paso 3: Configurar una función: LevelCurveTracings.m
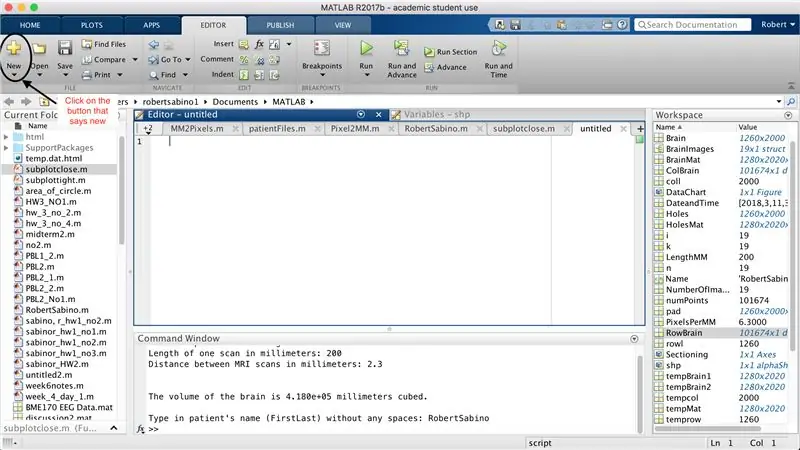
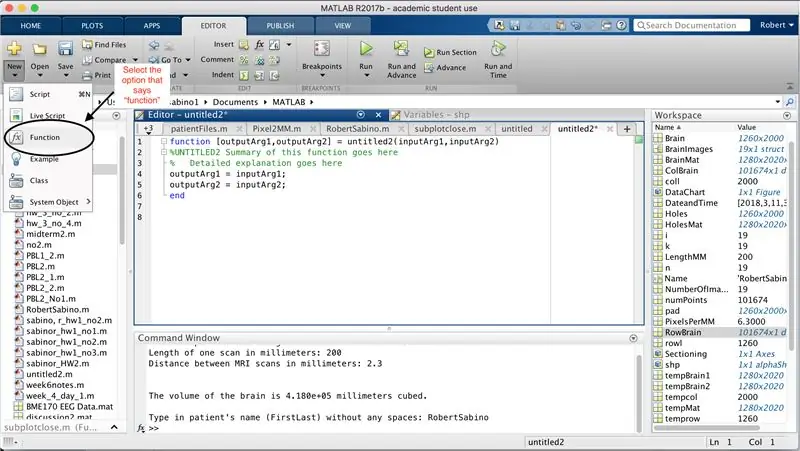
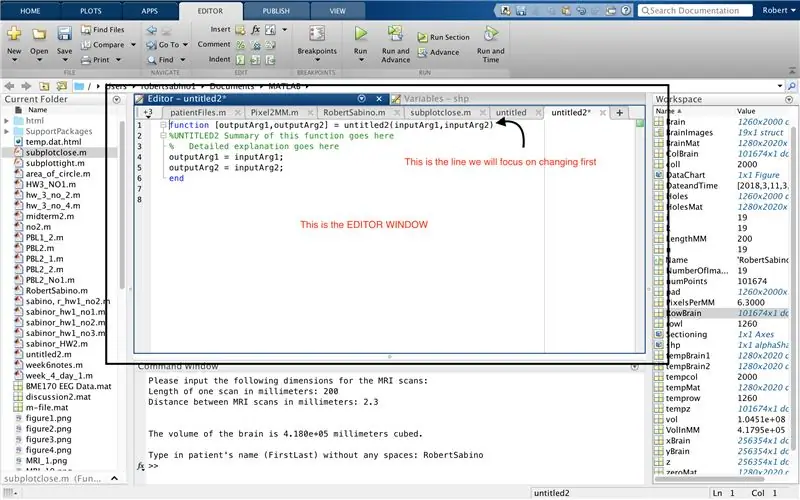

Primero, asegúrese de que su computadora tenga MATLAB_R2017b descargado (descargue aquí) y abra MATLAB. En la interfaz de MATLAB, haga clic en el botón en la esquina superior izquierda de la ventana que dice "Nuevo" con un signo más amarillo en negrita, y seleccione la opción "función", para abrir en la ventana del editor un espacio que se asemeja al de la tercera foto. Nos centraremos en cambiar la primera línea para configurar la función. Donde dice "outputArg1", reemplácelo con "brain", "outputArg2" para decir "agujeros", "untitled2" a "exp2" y "inputArg1" a "image", y elimine "inputArg2". Ahora tiene una función para ser llamada usando "exp2", tomando un argumento "imagen" y generando los límites de "cerebro" y "agujeros". La primera línea de la función debe parecerse a la línea que se muestra en la cuarta imagen. Elimine todo el código debajo de esta línea inicial.
Paso 4: Desarrollar el algoritmo de límites: encontrar límites

Escriba el código de la siguiente manera debajo de la línea. Esta sección de la función hace lo siguiente línea por línea.
- Cargue la imagen "imagen" en la variable "mri".
- Convierta "mri" en una imagen hecha de valores en un rango de números a unos y ceros (también conocido como binarización) basado en un valor de umbral establecido. Si el valor de un píxel es igual o superior a 0,1, se establece en uno; de lo contrario, el valor en ese píxel se establece en cero.
- Las siguientes cuatro líneas convierten 10 columnas y filas en los bordes de la capa de escaneo de MRI en ceros, para evitar leer valores incorrectos como límites (como se aprendió al experimentar con el código).
- En la última línea, bwbooulders traza los límites de la imagen binarizada "mri" y la iguala a "b", una matriz con los elementos cuyos índices corresponden a los de los límites establecidos en uno.
Paso 5: desarrollo del algoritmo enlazado: generación de una matriz enlazada externa
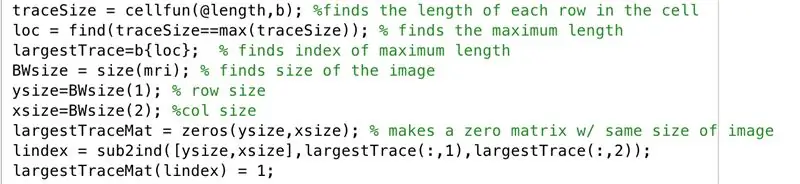
Siga en la ventana del editor con el siguiente código en la imagen. Esta sección del código hace lo siguiente línea por línea.
- Encuentre la longitud de cada una de las filas de la imagen binarizada "b" (cellfun aplica la longitud de la función a cada fila).
- Configure "loc" para almacenar las longitudes máximas.
- Encuentre el índice de la longitud máxima, configurado para almacenar en "largerTrace".
- Busque el tamaño de la imagen "mri", que tiene el mismo tamaño que "b", y configúrelo en "BWsize".
- Encuentre el número de filas en la matriz de la imagen, establecido en "ysize".
- Encuentre el número de columnas en la matriz de la imagen, establecido en "xsize".
- Genere la matriz "largerTraceMat", una matriz de ceros "ysize" por "xsize".
- Encuentre el índice equivalente a partir de los valores subindicados correspondientes a donde estaban los valores xy los valores y más grandes de Trace, almacénelos en el vector "lindex".
- En la matriz de ceros, "largerTraceMat", convierta los elementos en los índices que corresponden a los valores de índice almacenados como elementos en "lindex" en unos.
Por lo tanto, la matriz lógica "largerTraceMat" tiene la región acotada más grande de la sección transversal de exploración cerebral dada trazada como unos con un fondo de ceros.
Paso 6: Desarrollar el algoritmo delimitado: trabajar con el punto central

A continuación, debemos probar para ver si la sección transversal consta de más de una región (la más grande). Al probar la alineación del centroide de la región más grande, podemos ver si hay una región contigua, que produciría un centroide más centrado, o la posibilidad de múltiples regiones.
- Use "regionProps" para encontrar información sobre los centroides presentes, establezca igual a la matriz de estructura "tempStruct"
- Forme una matriz "centroides" con datos del campo "centroide" concatenados verticalmente
- Tome los valores de la segunda columna de "centroides" (las coordenadas de la dimensión horizontal)
- Ejecute un filtro para verificar la alineación del centroide con el centro horizontal
Paso 7: Desarrollo del algoritmo ligado: cuando un centroide no está centrado
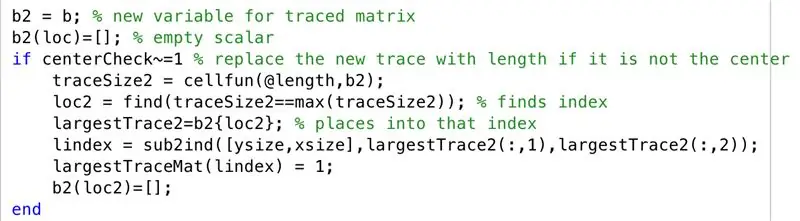
En el caso de que el centroide de la región de seguimiento más grande no esté centrado, seguimos los siguientes pasos. Como habíamos observado en las imágenes de resonancia magnética, la tendencia era tener hemisferios del cerebro representados en la sección transversal cuando no estaban contiguos, por lo que ahora continuamos trazando el segundo trazo más grande junto con el trazo más grande en "más grande TraceMat".
- Establecer la matriz rastreada en una nueva variable "b2"
- Inicializar la matriz vacía "b2", con un conjunto indexado por "loc"
- Cree un condicional, para cuando un centroide no está centrado (es decir, una capa de múltiples regiones)
- Establezca un nuevo tamaño de seguimiento para cada fila (traceSize2)
- Configure "loc2" para encontrar los índices donde están presentes los límites
- Deje que las celdas especificadas por "loc2" en "b2" sean iguales a "largerTrace2"
- Convierta los subíndices en índices, configúrelos en "lindex"
- Cambie los elementos correspondientes a "lindex" en "largerTraceMat" a 1
- Inicializar la matriz vacía "b2", con un conjunto indexado por "loc2"
Paso 8: Desarrollo del algoritmo ligado: interferencia de agujeros

Al tratar con los agujeros, los valores almacenados en "b2" mantuvieron un seguimiento de las estructuras distintas de la traza más grande, y graficarlas en una forma rellena de "más grande TraceMat" revelará dónde hay agujeros en las regiones del cerebro.
- Cree la matriz "fillMat", que es una forma completa de "largerTraceMat"
- Cree una matriz "interferenciaMat ", una matriz de ceros" ysize "por" xsize"
- Cree la matriz "interferencialoc ", para almacenar los valores de" b2 ", concatenados verticalmente
- Cree una matriz "lindex" para almacenar los índices que corresponden a "interferencialoc"
- Para los índices en "interferMat" que corresponden a "lindex", establezca el valor en 1, creando una región acotada diferente
Paso 9: Desarrollo del algoritmo delimitado: localización de los agujeros, finalización de los límites del cerebro y los límites del agujero

- Establezca la matriz "tempMat" igual a "interferenciaMat "más" fillMat ", sumando así cada valor en la matriz entre sí
- Establezca la matriz "holesLoc" igual a los índices donde "interferenciaMat "y" llenadoMat "eran iguales a uno
- Configure "holesMat" como una matriz cero de dimensiones "ysize" x "xsize"
- Establecer índices en "HoleMat" que sean iguales a "holesLoc" como unidades
- Establezca "cerebro" en "más grande TraceMat"
- Establecer "agujeros" en "HoleMat"
Con el hallazgo donde los valores de las matrices agregadas eran iguales a 2, las ubicaciones de los orificios se aseguraron fácilmente y se trazaron en una matriz vacía.
Paso 10: Registro de datos: Función PatientFiles.m
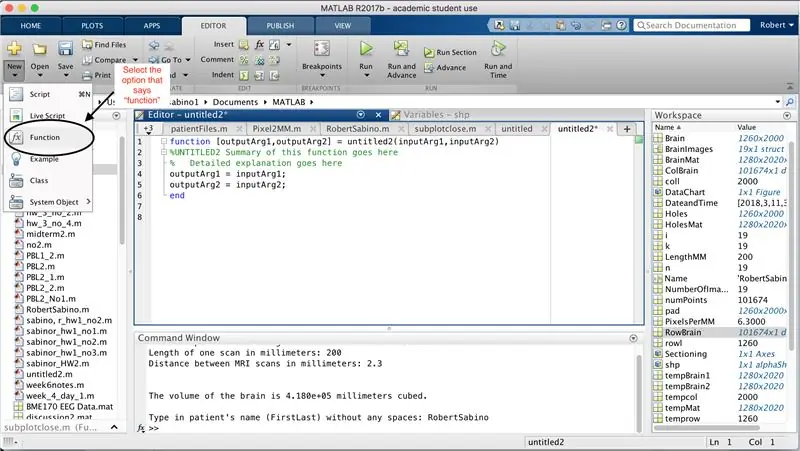
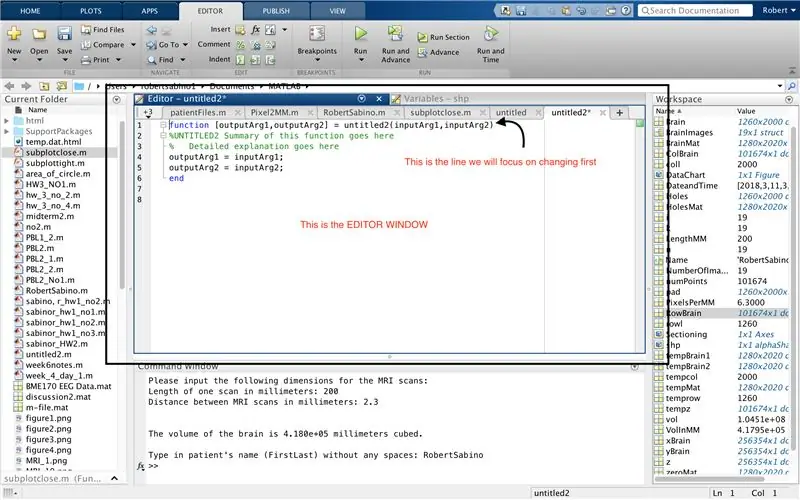

Al igual que la configuración de la última función, haga clic en el botón en la esquina superior izquierda de la ventana que dice "Nuevo" con un signo más amarillo en negrita, y seleccione la opción "función", para abrir en la ventana del editor un espacio que se parece al de la tercera imagen. En la primera línea, elimine la matriz de salida y reemplácela con simplemente "salida", reemplace "untitled2" con "PatientFiles", elimine todos los argumentos de entrada y, en su lugar, siga el formato especificado en la cuarta imagen de la línea de código. La primera línea de esta función debe coincidir con el formato de la imagen.
Paso 11: registrar datos en archivos
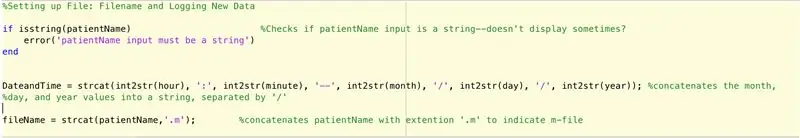

Para configurar un archivo para registrar los datos encontrados por la función principal (aún por describir), debemos seguir estos pasos (según lo prescrito por el código línea por línea).
- Compruebe si la entrada para el nombre del paciente es una cadena.
- Si no es una cadena, muestre que la entrada del nombre del paciente debe ser una cadena.
- Finaliza la sentencia if (evita errores).
- Configure una declaración de cadena "Fecha y hora" que dará el siguiente formato: hora: minutos - mes / día / año.
- Establezca la variable fileName en lo siguiente: PatientName.m.
Ahora a la siguiente sección de la función: ¿Existe ya un archivo con este nombre?
1) Suponga que el archivo con este nombre ya existe:
- Ejecute el archivo para obtener los valores del pasado en cola
- Agregue los datos "Fecha y hora" de la iteración actual como una nueva celda en la matriz de celdas de valores x (final del índice + 1)
- Agregue el valor actual de "brainVolume" como una nueva celda en la matriz de celdas de valores y (final del índice + 1)
- Guarde las variables actuales cargadas en el archivo.
2) Suponga que el archivo con este nombre no existe:
- Cree un nuevo archivo con el nombre almacenado en la variable "PatientName"
- Agregue los datos actuales de "Fecha y hora" como una celda en la matriz de celdas vacías de valores x
- Agregue los datos actuales de "brainVolume" como una celda en la matriz de celdas vacías de valores y
- Guarde las variables actuales cargadas en el archivo.
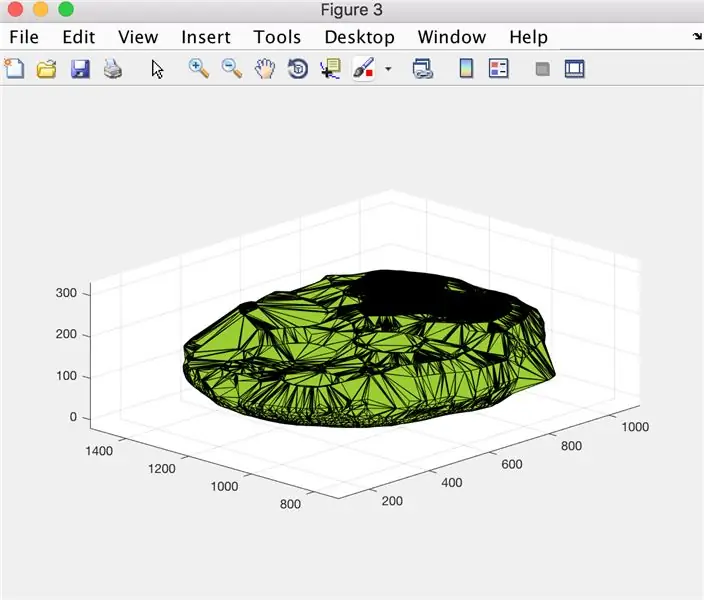
Paso 12: Registro de datos: visualización de un gráfico del volumen cerebral a lo largo del tiempo

- Convierta la matriz de valores x (xVals) en una matriz categórica (xValsCategorical), para permitir el trazado
- Generar ventana de figura 5
- Trace los puntos designados por "xValsCategorical" e "yVals" (que contienen el volumen del cerebro), utilizando círculos huecos para indicar puntos y para conectarlos mediante líneas discontinuas.
- Título de la gráfica como: nombre del paciente Datos de volumen cerebral
- Etiquete el eje x como se muestra en la imagen
- Etiquete el eje y como se muestra en la imagen
- Deje que la figura 5 sea igual a la salida
A partir de esto, la función PatientName que se llama generará un archivo con datos editados que mantienen un registro del volumen cerebral a lo largo del tiempo y un gráfico que muestra las tendencias.
Paso 13: Cerrar brechas en subparcelas: Subplotclose.m

La función, adaptada del código de https://www.briandalessandro.com, funciona para cerrar los espacios entre las figuras de la subparcela del código principal, cuando se crean las figuras que muestran las imágenes de resonancia magnética y las capas del cerebro. La función de subtrama utilizada dentro de subplotclose.m ajusta la posición de las subtramas dadas para encajar perfectamente entre sí en el aspecto de la dimensión más larga. Por ejemplo, si el código pretende una matriz de 7 x 3, las filas se ajustarán bien ya que la dimensión de la fila es más larga. Si el código tiene la intención de una matriz de 3 x 7, las columnas se ajustarán cómodamente, con espacios en las filas, como se muestra en las figuras de nuestro código principal.
Paso 14: El código principal: borrar todo y solicitar entradas

Para iniciar el código principal, haga clic en el mismo botón que dice "Nuevo" en la esquina superior izquierda de la ventana y seleccione "Script" en lugar de "Función" de las secciones anteriores. Escriba el código como se muestra en la imagen dentro de la ventana del editor. Las líneas de código realizan las siguientes tareas en orden:
- Cierre todos los archivos abiertos excepto 0, 1 y 2.
- Cierre todas las ventanas de figuras.
- Borre todas las variables en el espacio de trabajo.
- Limpia la ventana de comandos.
- Visualización en la ventana de comandos: ingrese las siguientes dimensiones para las exploraciones de resonancia magnética:
- En una nueva línea de la ventana de comandos, pregunte: Longitud de un escaneo en milímetros:. La respuesta introducida por el usuario se establecerá en la variable "lengthMM".
- En una nueva línea, pregunte: Distancia entre exploraciones de resonancia magnética en milímetros:. La respuesta introducida por el usuario se establecerá en la variable "ZStacks".
Paso 15: El código principal: procesamiento por lotes de las imágenes

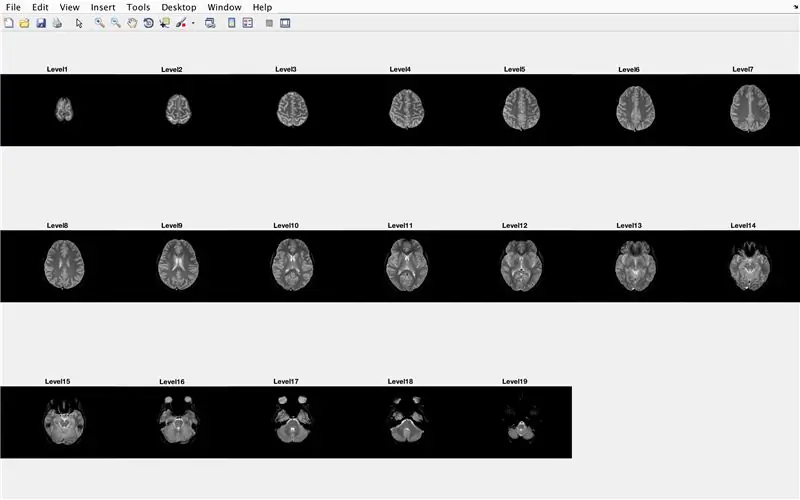
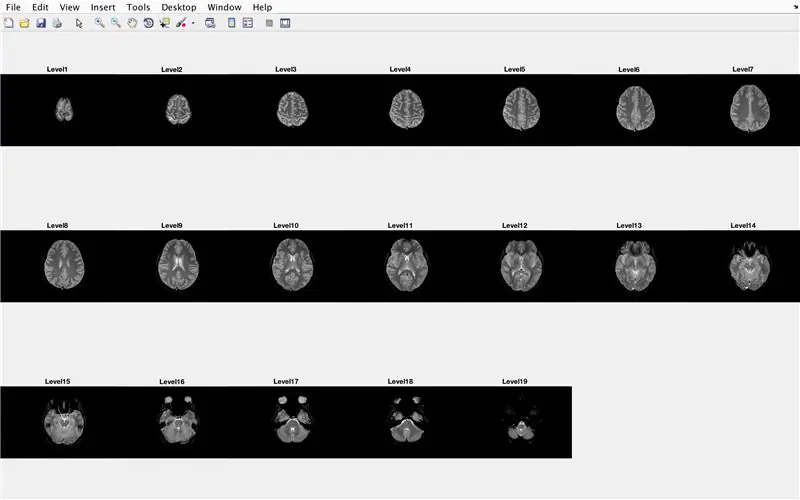
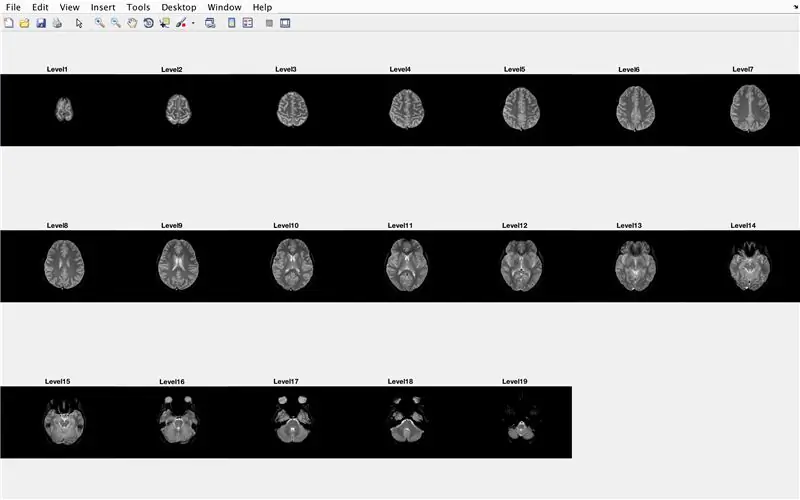
En esta sección, el código cargará las imágenes (que consisten en las imágenes por resonancia magnética de las secciones transversales del cerebro) y almacenará los nombres de cada archivo de imagen en la variable "Base" y mostrará cada una de las imágenes por resonancia magnética. Siga con el código de la imagen, que hace lo siguiente:
- Cree una matriz de estructura "BrainImages" que contenga información sobre todos los archivos dentro de la carpeta actual que se ajusten al formato de nombre de MRI _ (). Png
- Establezca la variable "NumberofImages" igual al número de elementos en la matriz de estructura "BrainImages"
- Ventana de figura abierta 1
- Configure un bucle for para recorrer el número de imágenes contadas en el archivo
- Para cada bucle, "CurrentImage" es el nombre respectivo de cada archivo MRI_i.png, con el número de iteración como 'i'
- Genere la subtrama de 3 x 7 para mostrar las 19 imágenes que se cargarán mediante "imshow"
- Mostrar cada imagen como otro elemento en la ventana de la figura de la subtrama
- Titula cada elemento de la subtrama como Nivel_, donde en blanco es el número de iteración del bucle for.
- Finalizar el ciclo for (evitando errores)
Esto mostrará en la ventana de la figura 1 todas las exploraciones de resonancia magnética en forma sin procesar en una configuración de 3 x 7 sin espacios en la orientación x.
Paso 16: El código principal: relleno

Con el relleno, evitamos el problema de las discrepancias leves en los tamaños de las imágenes que pueden producir un error por falta de coincidencia de dimensiones en el caso de que una imagen sea un poco más grande que otra.
- Ventana de figura abierta 2
- Cargue la matriz de imagen de MRI_1-p.webp" />
- Encuentre el tamaño de la matriz de la imagen y configúrelo en "OriginalXPixels" (para el número de filas) y "OriginalYPixels" (para el número de columnas)
- Configure la matriz "BrainMat" para que conste de todos los ceros con 20 filas más y 20 columnas más para cada plano, y 19 secciones transversales totales, una por plano.
- Configure "HolesMat" para que conste de la misma matriz tridimensional de ceros para ingresar las coordenadas del agujero más tarde
- Cree "zeroMat" para que tenga el tamaño de un pad más veinte filas y veinte columnas, una matriz bidimensional de ceros.
Paso 17: El código principal: Determinación de límites
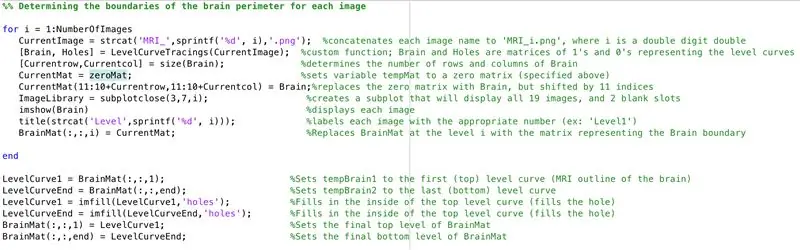
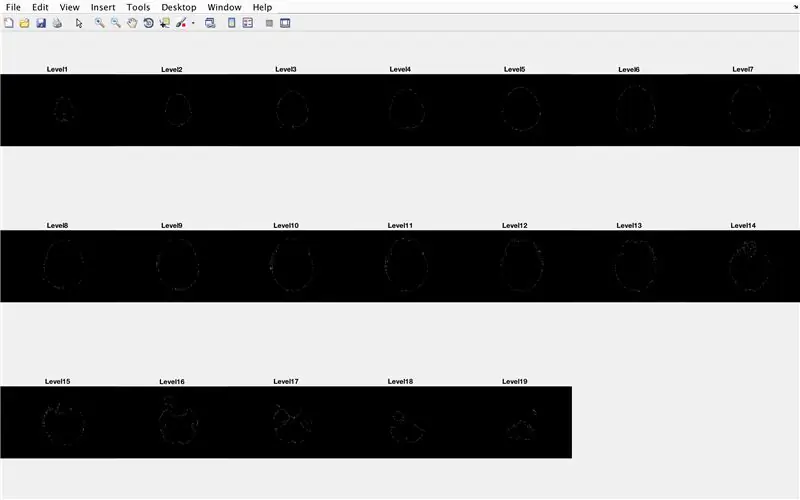
- Configure un bucle for para revisar los datos de cada imagen cargada anteriormente
- De la misma manera que antes se procesaba por lotes, "CurrentImage" carga archivos con "MRI_i.png", donde i es el número de iteración.
- Ejecute cada imagen a través de la función de procesamiento "LevelCurveTracings2.m" que creó anteriormente
- Encuentre el tamaño de la salida "Brain", establezca el número de filas en "Currentrow" y el número de columnas en "Currentcolumns"
- Establezca "CurrentMat" en una matriz de ceros con las dimensiones especificadas por "Currentrow" y "Currentcolumns"
- Centre los datos de "Brain" en "CurrentMat", con un margen de 10 filas en todos los lados
- Genere una subtrama de dimensiones 3 x 7, para mostrar los límites de las imágenes
- Título de cada uno de los elementos de la subtrama en la ventana de la figura
- Genere una matriz tridimensional "BrainMat" compuesta por cada capa de límites "CurrentMat"
- Finalizar el ciclo for (para evitar errores)
La subsección siguiente llena los agujeros que quedan en la parte superior e inferior de la forma tridimensional propuesta.
- Establezca "LevelCurve1" igual a la primera capa de "BrainMat" (parte inferior del sólido)
- Establezca "LevelCurveEnd" igual a la capa final de "BrainMat" (parte superior del sólido)
- Sobrescribir "LevelCurve1" con una capa rellena
- Sobrescribir "LevelCurveEnd" con una capa rellena
- Establecer la capa rellena como la capa inferior de "BrainMat"
- Establecer la capa rellena como la capa superior de "BrainMat"
Paso 18: El código principal: determinar la dimensión Z adecuada

Las primeras tres líneas consisten en configurar una matriz vacía "z" y realizar operaciones de conversión simples (dividir píxeles por longitud) para obtener una lectura adecuada del volumen en mm ^ 3
- Crea un bucle for para recorrer cada capa
- Encuentra el número de unos en una capa determinada
- Convierta las coordenadas z de las unidades en valores que se escalen a la proporción adecuada, establecidos en "tempz", un vector de columna
- Agregue el valor z para la curva de nivel al vector z
Con esto las coordenadas z se ajustan correctamente.
Paso 19: El código principal: determinación de las coordenadas X e Y

Ahora para determinar las posiciones xey de cada uno de los puntos en los límites.
- Inicializar "xBrain" como una matriz vacía
- Inicializar "yBrain" como una matriz vacía
- Configure un bucle for para recorrer cada imagen cargada
- Compile una matriz de dos columnas para almacenar las coordenadas planas de cada punto en el límite, representadas por los vectores de columna "RowBrain" y "ColumnBrain"
- Agrega "xBrain" con las coordenadas "RowBrain" encontradas actualmente
- Agrega "yBrain" con las coordenadas "ColumnBrain" encontradas actualmente
- Finalizar el ciclo for (para evitar errores)
Paso 20: El código principal: trazar una estructura tridimensional, encontrar volumen y registrar datos
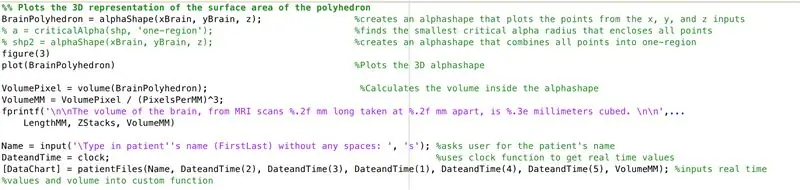

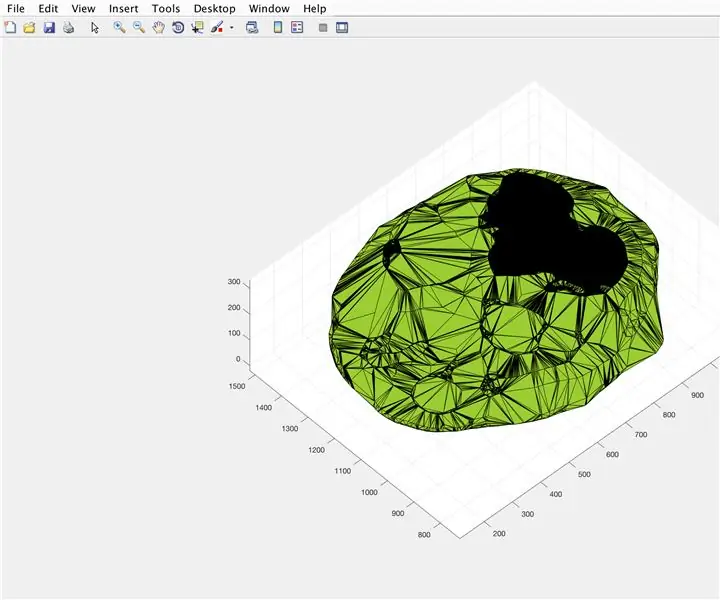
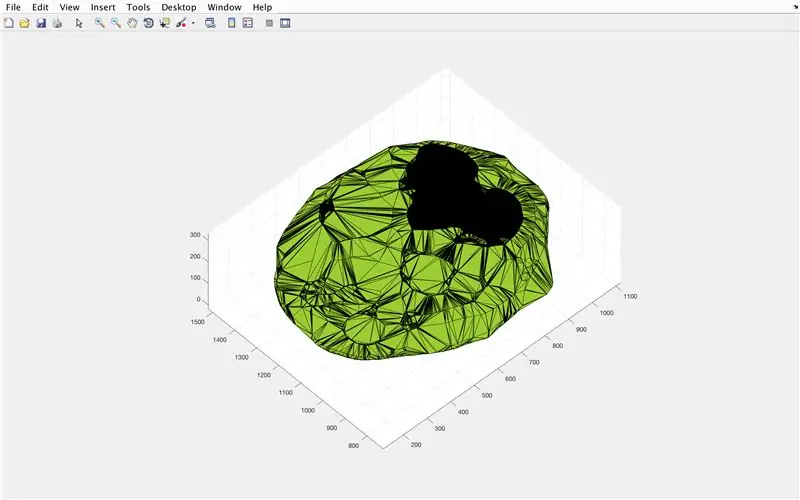
Usando la función alphaShape, crearemos una estructura tridimensional a partir de la cual podemos calcular el volumen del cerebro.
- Utilice la función alphaShape, inserte los vectores "xBrain", "yBrain" y "z" para las coordenadas x, y y z, y establezca el valor "BrainPolyhedron"
- Ventana de figura abierta 3
- Trazar la forma alfa calculada "BrainPolyhedron", mostrar en la ventana de la figura
- Calcule el volumen de la forma alfa mediante una función "volumen" que funcione para formas alfa
- Convierta el volumen en mm ^ 3
- Imprima el volumen del sólido en la ventana de comandos
- Solicitar que se establezca un nombre de paciente como entrada
- Obtener la fecha y la hora actuales con el reloj y configurarla en "Fecha y hora"
- Llame a la función "PatientFiles" para registrar y trazar los datos calculados
A partir de aquí, la segunda y tercera imágenes deben mostrar las figuras que aparecen, y la cuarta imagen la que debe mostrarse en la ventana de comandos.
Recomendado:
Kits de coche de seguimiento de robot inteligente de bricolaje Seguimiento de coche fotosensible: 7 pasos

DIY Smart Robot Tracking Car Kits Tracking Car Photosensitive: Design by SINONING ROBOTPuedes comprar desde el robot de seguimiento carTheoryLM393 chip compara los dos fotorresistores, cuando hay un fotorresistor LED en BLANCO, el lado del motor se detendrá inmediatamente, el otro lado del motor girar, de modo que
Time Cube - Gadget de seguimiento de tiempo de Arduino: 6 pasos

Time Cube - Arduino Time Tracking Gadget: Me gustaría proponerle un proyecto arduino simple pero realmente útil para rastrear eventos de tiempo girando algún gadget de cubo inteligente. Dale la vuelta a " Trabajo " > " Aprender " > " Tareas " > " Descanso " lado y contará el
Seguimiento GPS en tiempo real: 3 pasos
Seguimiento GPS en tiempo real: en este tutorial, le mostraremos cómo realizar un seguimiento de la ubicación en tiempo real utilizando un BerryGPS-GSM, un Raspberry Pi Zero y un estado inicial. Enviaremos longitud, latitud y velocidad a través de 3G con el BerryGPS-GSM al estado inicial
Cómo iluminar una perilla de volumen del receptor de audio y video. (onkyo Hr550): 3 pasos

Cómo iluminar una perilla de volumen del receptor de audio y video. (onkyo Hr550): las perillas de volumen retroiluminadas son algo así como una creación reciente. Realmente no tiene ninguna funcionalidad, pero se ve muy bien. Conseguí un receptor HR550 para Navidad y decidí lanzar un tutorial sobre cómo hacer esto. Elementos necesarios: Soldador multímetro
¡Rueda de desplazamiento del escritorio y control de volumen !: 8 pasos (con imágenes)

¡Rueda de desplazamiento del escritorio y control de volumen !: Proyecto de fin de semana de Make Magazine, 4/3/09 Algunos antecedentes Recientemente, he estado leyendo muchos blogs diferentes. Cada día leeré las últimas publicaciones sobre engadget, lifehacker, hackaday, BBG y el blog MAKE :. El problema para mí vino cuando me di cuenta de que o
