Tabla de contenido:
- Paso 1: Organización de los datos de la mamografía
- Paso 2: procesamiento de imágenes
- Paso 3: Código de umbral
- Paso 4: encontrar anomalías para cada imagen binaria
- Paso 5: Trazar la ubicación masiva y el tamaño diagnosticados para la comparación visual
- Paso 6: Implementación del segundo método de comparación
- Paso 7: análisis de los datos recopilados
- Paso 8: ¡Haga su propio clasificador
- Paso 9: ¿Mejoras? ¿Alguna idea?
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 08:42.
- Última modificación 2025-01-23 14:39.
El propósito de este proyecto era identificar y utilizar un parámetro para procesar imágenes de mamografías en escala de grises de varias clasificaciones de tejido de fondo: tejido graso, glandular graso y tejido denso. Esta clasificación se utiliza cuando los radiólogos analizan mamografías y deben considerar si la densidad del tejido ocultará cualquier anomalía, como lesiones o tumores. Esto se debe tanto a las estructuras fisiológicas normales como el tejido glandular y el tejido conectivo fibroso. y las morfologías anormales, como calcificaciones y tumores, aparecerán muy brillantes en la mamografía, mientras que el tejido graso menos denso aparecerá de color negro. Por lo tanto, fue apropiado programar un clasificador que pueda manipular los niveles de intensidad de píxeles para visualizar e identificar mejor las masas.
Paso 1: Organización de los datos de la mamografía
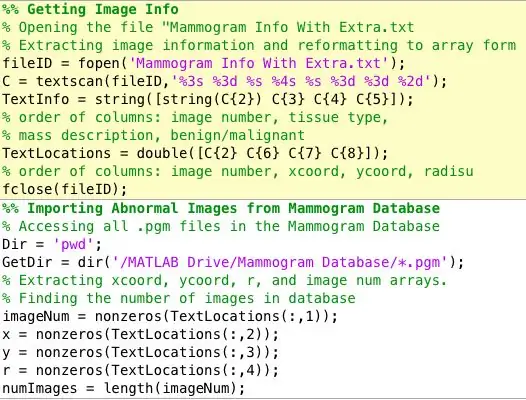
Una de las primeras cosas que me di cuenta de que necesitaba manejar fue organizar los datos de una manera muy clara, concisa y accesible. Estas son las variables que extraje de la base de datos de mamografías mini-MIAS. Creé dos matrices. Uno que contiene 4 columnas:
- Número de imagen:
- x coordenada de masa
- y coordenada de masa
- Radio de masa: (Esto definió un tamaño aproximado para la masa
La segunda matriz contenía información de clasificación:
- Tipo de tejido de fondo: graso (F), glandular graso (G), denso (D)
- Descripción de la masa: bien definida (CIRC), espiculada (SPIC), otra mal definida (MISC) Distorsión arquitectónica (ARCH), asimetría (ASYM), normal (NORM)
- Diagnósticos: benigno (B), maligno (M)
Dado que el objetivo de este proyecto era determinar el mejor umbral para cada tipo de tejido de fondo, no se necesitaba toda la información. Sin embargo, puede expandir su proyecto para incluir análisis de textura y probar su clasificador contra las descripciones de masas conocidas.
Nota al margen: La base de datos de la que obtuve las imágenes de mamografía diagnosticadas organizó la información sobre cada mamografía en un archivo de texto separado de las imágenes. Fue un poco difícil para mí extraer los datos de un archivo de texto y organizarlos en formas de matriz, pero el siguiente enlace fue muy útil para resolver todo eso. Alternativamente, simplemente ajuste el código que pegué arriba para sus propósitos.
Formato de archivo de mamografía: mdb001 G CIRC B 535425197
mdb002 G CIRC B 522280 69
Ayuda de TextScan: https://www.mathworks.com/help/matlab/ref/textsca… Base de datos de mammogramas:
Paso 2: procesamiento de imágenes
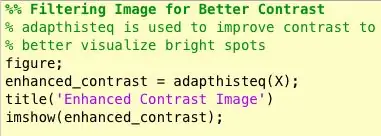
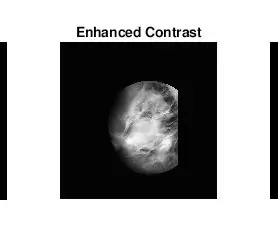
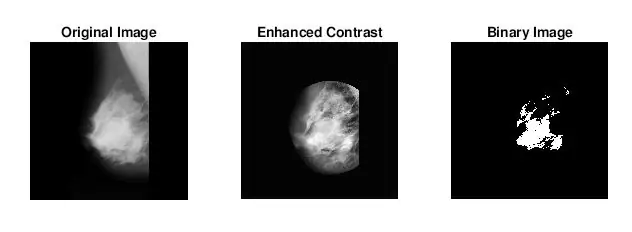
Bueno, lo segundo que surgió cuando estaba descubriendo cómo identificar masas fue que para muchas mamografías anormales no podía decir visualmente dónde estaba la anormalidad o qué tan grande era. Obviamente, como no soy un radiólogo experimentado, era de esperar. Sin embargo, la forma más sencilla de encontrar anomalías (según mis largas búsquedas en Google) era observar concentraciones de áreas brillantes y oscuras. Usé principalmente la función adapthisteq para mejorar el contraste de la imagen y luego imbinarizar para convertir la imagen en una imagen binaria para experimentar con diferentes niveles de umbral.
- adapthisteq: esta función transforma los valores de intensidad de las imágenes en escala de grises y rgb utilizando la ecualización de histograma adaptativo limitado por contraste. En otras palabras, ajusta el histograma de valores de intensidad a un tipo específico de distribución. El enlace de Mathworks para esta función se adjunta a continuación para leer más.
- imbinarize: crea una imagen binaria a partir de una imagen en escala de grises asignando todos los píxeles por encima de una determinada intensidad a 1 y los píxeles por debajo de ese valor un 0. Usé esta función para probar el umbral óptimo para reducir el ruido del tejido de fondo.
Paso 3: Código de umbral
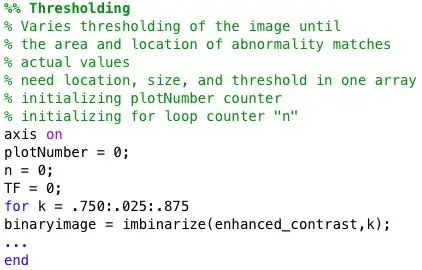
Se utiliza un bucle for para binarizar la mamografía con distintos umbrales. Para ofrecer una imagen más amplia, el bucle for contiene el código del Paso 3 al Paso 7. Por lo tanto, cada imagen binaria se analizará en busca de anomalías. Además, este bucle for está incluido en otro bucle for que importa una nueva imagen de mamografía de la base de datos en cada iteración.
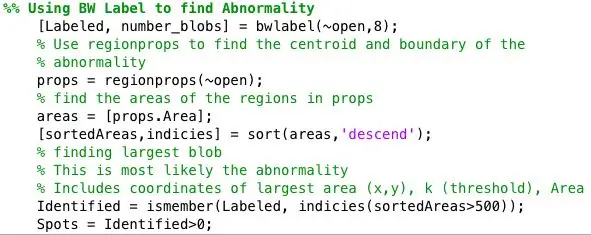
Paso 4: encontrar anomalías para cada imagen binaria
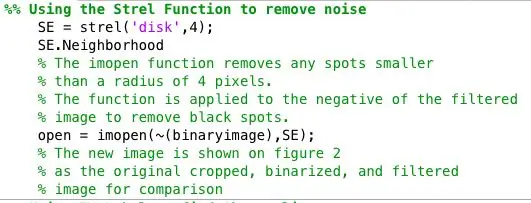
Procesé aún más las imágenes binarias usando la función strel junto con imopen para eliminar el ruido de fondo. La imagen binaria del paso anterior se invierte y se filtra utilizando la vecindad definida por SE. Luego usé bwlabel para etiquetar cualquier área que tuviera al menos 8 píxeles conectados.
La función de apoyos de la región se utilizó para encontrar el centroide y las propiedades del área de cada punto identificado por bwlabel.
Luego, todos los puntos de más de 500 píxeles se identificaron usando ismember. Los centroides para los puntos identificados se trazaron en una imagen que solo mostraba los puntos con un área mayor a 500. Área identificada = ismember (Etiquetado, indicies (sortedAreas> 500)); Manchas = Identificado> 0;
Paso 5: Trazar la ubicación masiva y el tamaño diagnosticados para la comparación visual
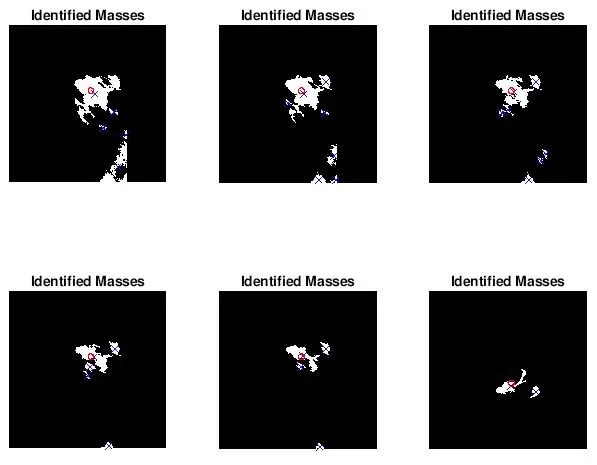
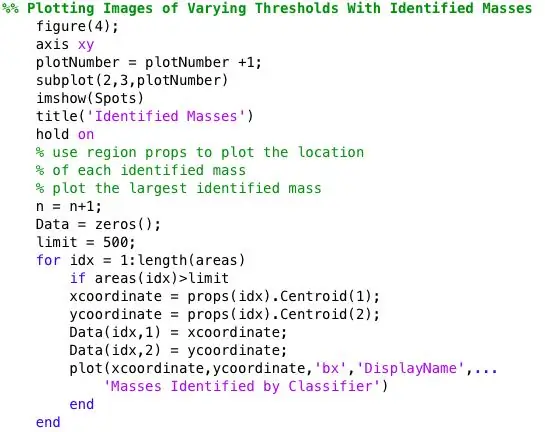

Quería ver si las manchas encontradas por bwlabel eran correctas. Hice esto de dos formas. Primero analicé la precisión de mi clasificador haciendo una comparación visual. Simplemente tracé el tamaño real y la ubicación de la anomalía (círculo rojo) y la ubicación determinada por el código (x azul) en la imagen de la mamografía preprocesada. Las seis imágenes de arriba muestran los efectos de aumentar el valor de umbral de la escala de grises.
Paso 6: Implementación del segundo método de comparación

La segunda forma en que probé el clasificador y los valores de umbral fue determinando si las ubicaciones encontradas por el clasificador estaban dentro de una cierta distancia de las coordenadas de la anomalía diagnosticada. Guardé los umbrales para los cuales al menos uno de los puntos identificados estaba dentro de 1,5 * r de la anomalía conocida en un archivo de texto separado llamado Mammogram Data. El propósito de esto era encontrar el umbral mínimo necesario para que mi clasificador identificara la anomalía.
Paso 7: análisis de los datos recopilados
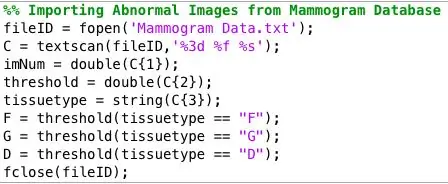
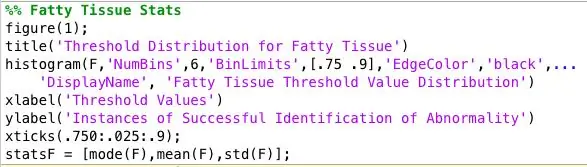
Ejecuté el programa en todas las imágenes de mamografías anormales y me quedé con un archivo de texto enorme de datos. Para encontrar el mejor umbral para cada tipo de tejido, organicé los datos por tipo de tejido y tracé un histograma de los valores de umbral para cada tipo de tejido. Se decidió el valor de umbral adecuado sobre qué umbral proporcionaba los resultados más precisos para cada tipo de tejido. Guardé estos datos para subirlos a mi clasificador.
Paso 8: ¡Haga su propio clasificador

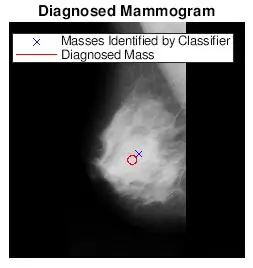
Después de encontrar los valores de umbral más apropiados para cada tipo de tejido, edité mi código original para que un usuario ingresara el número de imagen y el tipo de tejido para elegir el umbral para la imagen de la mamografía. Luego tracé la ubicación de la mamografía diagnosticada con las ubicaciones encontradas en las imágenes de la mamografía original. Quería hacer esto más divertido, así que programé una función para recortar una región circular que rodea el ROI. Se le indicará al usuario que elija un punto central y varios puntos que abarquen mejor el ROI. Adjunté ambos archivos de matlab aquí.
Paso 9: ¿Mejoras? ¿Alguna idea?
Mientras escribía este instructivo, comencé a ver muchas mejoras que podría hacer al clasificador, como encontrar formas de distinguir entre los diferentes tipos de masas identificadas en función del análisis de textura o mejorar mi sección de pruebas de precisión del SandBoxProject. expediente. Como se trataba de un proyecto con una fecha límite, tuve que detenerme en algún lugar, pero espero poder utilizar las habilidades de procesamiento de imágenes que aprendí en otras aplicaciones. Además, adjunté el archivo que se utilizó para procesar por lotes todas las imágenes de mamografías anormales.
Recomendado:
Intensidad de la señal WiFi ESP32 TTGO: 8 pasos (con imágenes)

Intensidad de la señal WiFi ESP32 TTGO: En este tutorial aprenderemos cómo mostrar la intensidad de la señal de una red WiFi usando la placa ESP32 TTGO. ¡Mira el video
Medidor de intensidad de luz sin programación: 7 pasos (con imágenes)

Medidor de intensidad de luz sin programación: este instructivo trata de hacer un medidor de intensidad de luz básico sin usar Arduino o cualquier otro microcontrolador o programación. El medidor de intensidad de luz muestra diferentes niveles de intensidad de luz con diferentes colores de LED. El LED rojo
Circuito de luz de vela OLED con fotorresistencia para control de intensidad (TfCD): 4 pasos (con imágenes)

Circuito de luz de vela OLED con fotoresistencia para control de intensidad (TfCD): en este instructivo le mostramos cómo hacer un circuito que muestre (O) LED parpadeando como una vela y reaccione a la intensidad del ambiente. Con una intensidad de luz más baja, se necesita una salida de luz más baja de las fuentes de luz. Con esta aplicación
N: Cómo hacer una escultura de acrílico y LED de varias capas con niveles de iluminación variables: 11 pasos (con imágenes)

N: Cómo hacer una escultura de acrílico y LED de varias capas con niveles de iluminación variables: aquí puede descubrir cómo hacer su propia n como hecha para la exposición www.laplandscape.co.uk curada por el grupo de arte / diseño Lapland. Se pueden ver más imágenes en flickr. Esta exposición estará abierta desde el miércoles 26 de noviembre hasta el viernes 12 de diciembre de 2008 inclusive
Estéreo para automóvil con alimentación ATX y altavoces de 3 vías (para uso doméstico): 10 pasos (con imágenes)

Estéreo para automóvil con alimentación ATX y parlantes de 3 vías (para uso doméstico): Ha pasado un tiempo desde que investigué sobre cómo encender el estéreo de un automóvil sin una batería de 12 voltios que seguramente tendré que recargar más tarde. ¿Por qué? bueno … porque tengo una unidad sony mp3 cd usb aux ipod-cable, 4x52w watts w / sub-out, qué más
