Tabla de contenido:
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 08:40.
- Última modificación 2025-01-23 14:39.
Por: Phuc Lam, Paul Yeung, Eric Reyes
Reconocer que los errores en la segmentación de los pulmones producirán información falsa con respecto a la identificación de un área patológica y pueden afectar directamente el proceso de diagnóstico. Las técnicas modernas de ayuda informática no lograron obtener resultados precisos cuando las enfermedades pulmonares tienen formas desafiantes. Estas formas anormales pueden ser causadas por derrames pleurales, consolidaciones, etc. Al aplicar la técnica de segmentación pulmonar, en la que los límites del pulmón se aíslan del tejido torácico circundante, nuestra aplicación puede identificar los límites con los umbrales de entrada del usuario para brindar vistas totalmente personalizables. de las formas de los pulmones, El propósito de este proyecto MatLab es crear una aplicación interactiva de segmentación pulmonar fácil de usar para detectar condiciones patológicas de las imágenes de rayos X de los pulmones. Nuestro objetivo es crear una forma más eficaz de ilustrar e identificar pulmones anormales a fin de ofrecer a los médicos y radiólogos una forma más fiable de diagnosticar enfermedades pulmonares. Usando la herramienta de diseño de aplicaciones en MatLab, el programa está diseñado para funcionar específicamente con radiografías de tórax y tomografías computarizadas (TC), pero también está probado para funcionar con imágenes por resonancia magnética.
Las instrucciones a continuación contienen nuestra técnica de filtrado de ruido (filtro Wiener de paso bajo), así como el umbral de la imagen (usando el histograma de intensidad de la imagen en escala de grises) y usando un gradiente morfológico (la diferencia entre la dilatación y la erosión de una imagen) para identificar una región de interés. Luego, la instrucción explicará cómo integramos todos los elementos en la interfaz gráfica de usuario (GUI).
Nota:
1). Este proyecto está inspirado en un trabajo de investigación: "Segmentación y análisis de imágenes de pulmones anormales en CT: enfoques actuales, desafíos y tendencias futuras". Que se puede encontrar aquí
2). Estamos utilizando imágenes de rayos X de NIH: Clinical Center. El enlace se puede encontrar aquí
3). La ayuda del diseñador de aplicaciones se puede encontrar aquí
4). Antes de ejecutar el código: debe cambiar la ruta Dir (en la línea 34) a su directorio de archivos y el tipo de imagen (línea 35) (estamos analizando *.png).
Paso 1: Paso 1: carga de la imagen
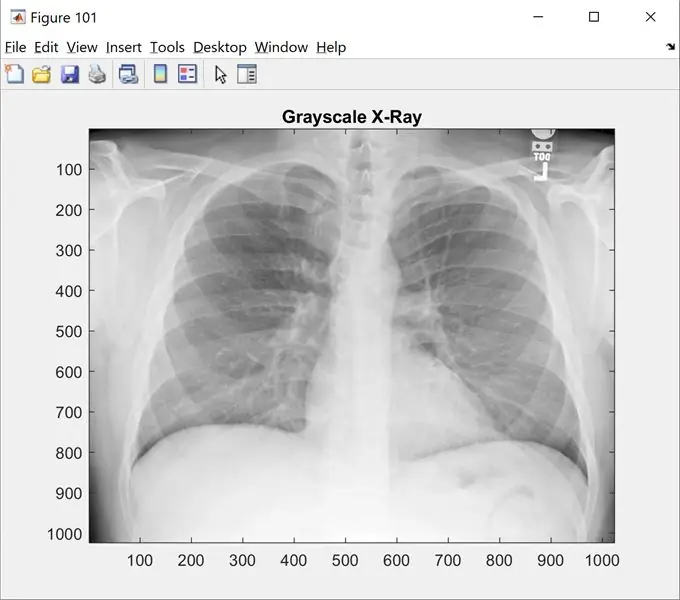
Este paso le mostrará la imagen original en escala de grises. Cambie 'name_of_picture.png' por el nombre de su imagen
claro; clc; cierra todo;
%% Cargando imágenes
raw_x_ray = 'nombre_de_imagen.png';
I = imread (raw_x_ray);
figura (101);
imshow (yo);
mapa de colores (gris);
título ('Rayos X en escala de grises');
Paso 2: Paso 2: Filtrado de ruido e histograma
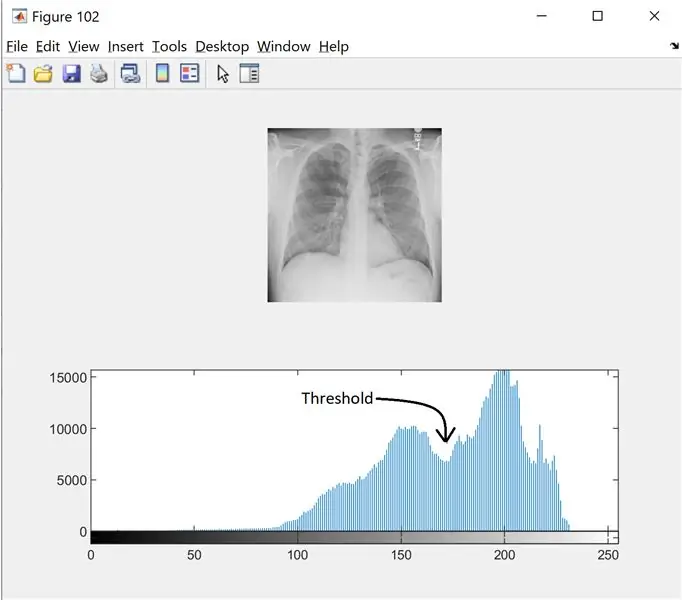
Para encontrar el umbral de la imagen en escala de grises, miramos el histograma para ver si hay modos distintos. Leer más aquí
I = wiener2 (I, [5 5]);
figura (102);
subtrama (2, 1, 1);
imshow (yo);
subtrama (2, 1, 2);
imhist (I, 256);
Paso 3: Paso 3: Establecer umbrales
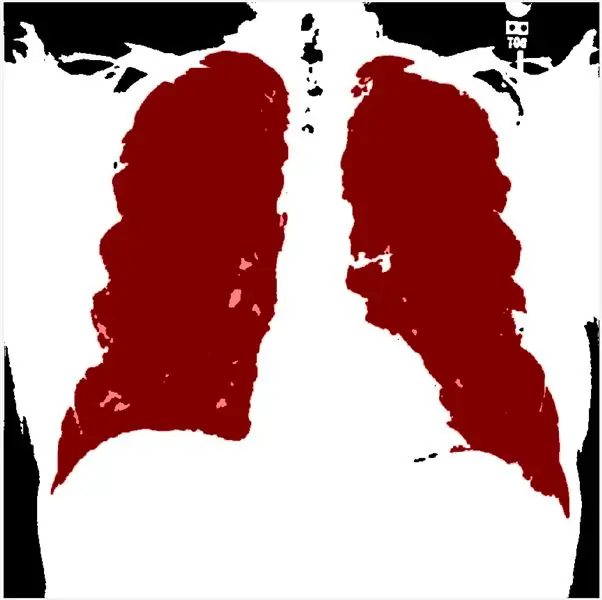
Este paso le permite establecer el umbral de acuerdo con el histograma. morphologicalGradient resaltará la región de interés en rojo, y la función visboundary se superpone a la imagen delineada y filtrada del pulmón en rojo.
Mediante el uso de regionprops, podemos obtener las matrices de solidez y ordenarlas de forma descendente. A continuación, binarizo la imagen de sclae gris y aplico el método de gradiente morfológico y mLoren Shurasking para resaltar la región de interés (ROI). El siguiente paso es invertir la imagen para que el ROI del pulmón sea blanco sobre fondo negro. Utilizo la función showMaskAsOverlay para mostrar 2 máscaras. Nota: el código está inspirado en Loren Shure, enlace.
Por último, creo un contorno rojo usando los límites bwbw y enmascarando la imagen del filtro y los límites.
a_thresh = I> = 172; % establece este umbral
[labelImage, numberOfBlobs] = bwlabel (a_thresh);
props = regionprops (a_thresh, 'todos');
sortedSolidity = sort ([props. Solidity], 'descender');
SB = sortedSolidity (1);
si SB == 1% SB solo acepta solidez == 1 filtra los huesos
binaryImage = imbinarize (I); figura (103);
imshow (binaryImage); mapa de colores (gris);
SE = strel ('cuadrado', 3);
morphologicalGradient = imsubtract (imdilate (binaryImage, SE), imerode (binaryImage, SE));
mask = imbinarize (morphologicalGradient, 0.03);
SE = strel ('cuadrado', 2);
máscara = imclose (máscara, SE);
mask = imfill (máscara, 'agujeros');
mask = bwareafilt (máscara, 2); % de control del número de área mostrada
notMask = ~ máscara;
máscara = máscara | bwpropfilt (notMask, 'Área', [-Inf, 5000 - eps (5000)]);
showMaskAsOverlay (0.5, máscara, 'r'); % tienes que descargar la aplicación / función showMaskAsOverlay
BW2 = imfill (binaryImage, 'agujeros');
nueva_imagen = BW2;
nueva_imagen (~ máscara) = 0; % fondo invertido y agujeros
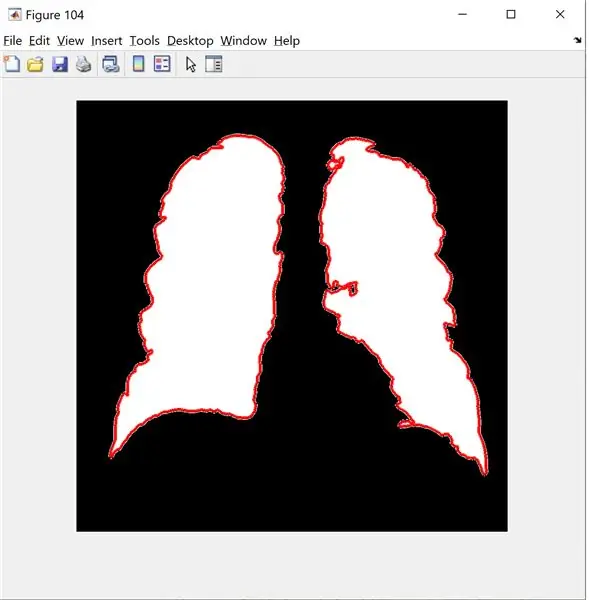
B = bwboundary (nueva_imagen); % solo puede aceptar 2 dimensiones
figura (104);
imshow (nueva_imagen);
esperar
límites fronterizos (B);
fin
Paso 4: creación de GUI
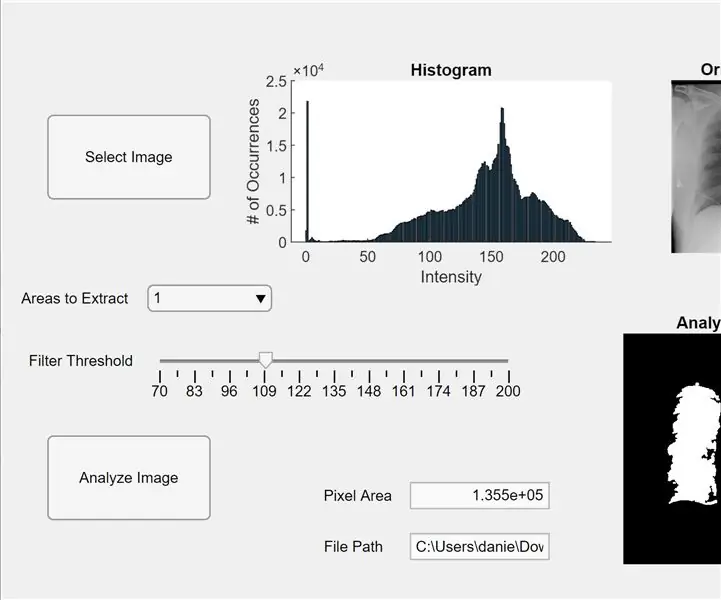
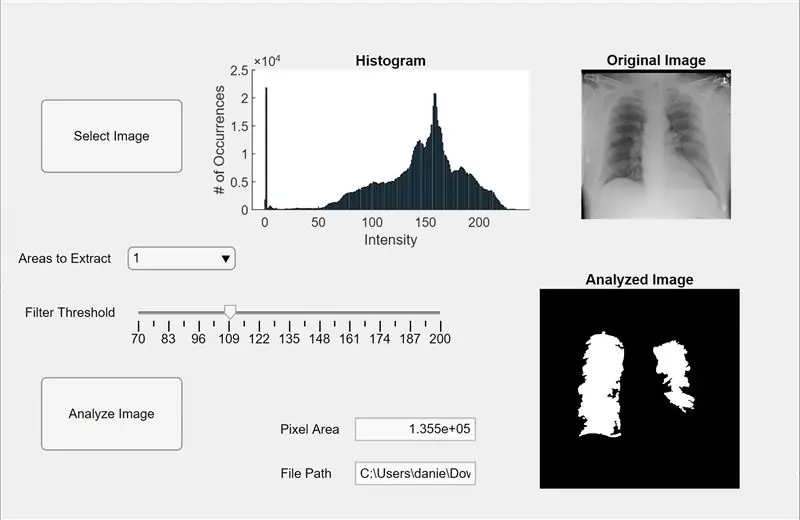
Ahora, integramos el código anterior en una aplicación MATLAB. Abra el Diseñador de aplicaciones en MATLAB (Nuevo> Aplicación). Primero, diseñamos la interfaz haciendo clic, manteniendo presionado y arrastrando tres ejes al espacio de trabajo central. A continuación, hacemos clic, mantén presionado y arrastramos dos botones, un campo de edición (texto), un campo de edición (numérico), un control deslizante y un menú desplegable. Dos ejes mostrarán cada uno la vista previa y analizarán la imagen, y el tercer eje mostrará un histograma de píxeles para la imagen "seleccionada" de vista previa. El cuadro del campo de edición (texto) mostrará la ruta del archivo de la imagen seleccionada y el campo de edición (numérico) mostrará el área de píxeles detectada de los pulmones.
Ahora cambie de la vista de diseño a la vista de código en App Designer. Introduzca en el código el código de las propiedades haciendo clic en el botón rojo "Propiedades" con un signo más. Inicialice las propiedades I, el umbral y las regiones para extraer como en el código que se proporciona a continuación. A continuación, haga clic con el botón derecho en un botón en la parte superior derecha del espacio de trabajo (el Explorador de componentes) y vaya a Devolución de llamada> Ir a … devolución de llamada. Agregue el código para la "función SelectImageButtonPushed (aplicación, evento)". Este código le permite seleccionar una imagen para analizar desde su computadora usando uigetfile. Después de seleccionar una imagen, aparecerá una imagen de vista previa debajo de los ejes acompañada de un histograma. Luego, haga clic con el botón derecho en el otro botón y repita el mismo procedimiento para crear una función de devolución de llamada.
Agregue el código debajo de la "función AnalyzeImageButtonPushed (aplicación, evento)". Este código realizará el recuento de píxeles y la detección de manchas en la imagen de vista previa en el botón analizar imagen (cualquiera en el que haya hecho clic con el botón derecho para este código). Después de programar los botones, ahora programaremos el control deslizante y el menú desplegable. Haga clic con el botón derecho en el control deslizante, cree una función de devolución de llamada y agregue el código debajo de "función FilterThresholdSliderValueChanged (aplicación, evento)" hasta el final. Esto permite que el control deslizante ajuste el umbral de intensidad de gris.
Cree una función de devolución de llamada para el menú desplegable y agregue el código debajo de "función AreastoExtractDropDownValueChanged (aplicación, evento)" para permitir que el menú desplegable modifique el número de manchas que se muestran en los ejes de la imagen analizada. Ahora, haga clic en cada entidad en el Explorador de componentes y cambie sus propiedades a su gusto, como cambiar los nombres de las entidades, eliminar ejes y cambiar la escala. Arrastre y suelte las entidades del Explorador de componentes en la vista Diseño a un diseño funcional y fácil de entender. ¡Ahora tiene una aplicación en MATLAB que puede analizar imágenes de pulmones para el área de píxeles!
propiedades (Acceso = privado) I = ; % archivo de imagen
umbral = 257; % de umbral para binarizar la intensidad del gris
RegionsToExtract = 2;
fin
función SelectImageButtonPushed (aplicación, evento)
clc; Dir = 'C: / Usuarios / danie / Descargas / images_004 / images'; % define archivo invariable "prefijo"
[imageExt, ruta] = uigetfile ('*. png'); % agarra la parte variable del nombre de la imagen
imageName = [Dir filesep imageExt]; % concatenar extracciones invariables y variables
app. I = imread (nombre de la imagen); % lee la imagen
imshow (aplicación I, 'padre', aplicación. UIAxes); % muestra la imagen
app. FilePathEditField. Value = ruta; % muestra la ruta del archivo de donde proviene la imagen original
fin
función AnalyzeImageButtonPushed (aplicación, evento)
originalImage = app. I;
Imagen original = wiener2 (aplicación. I, [5 5]); % filtro de eliminación de puntos
histograma (aplicación AxesHistogram, aplicación I, 256); % muestra el histograma de la imagen
a_thresh = originalImage> = app.threshold; % establece este umbral
labelImage = bwlabel (a_thresh);
props = regionprops (a_thresh, 'todos');
sortedSolidity = sort ([props. Solidity], 'descender');
SB = sortedSolidity (1);
si SB == 1% SB solo acepta solidez == 1 filtra los huesos
SE = strel ('cuadrado', 3);
morphologicalGradient = imsubtract (imdilate (labelImage, SE), imerode (labelImage, SE));
mask = imbinarize (morphologicalGradient, 0.03);
SE = strel ('cuadrado', 2);
máscara = imclose (máscara, SE);
mask = imfill (máscara, 'agujeros');
mask = bwareafilt (máscara, app.regionsToExtract);
% de control del número de área mostrada
notMask = ~ máscara;
máscara = máscara | bwpropfilt (notMask, 'Área', [-Inf, 5000 - eps (5000)]);
BW2 = imfill (labelImage, 'agujeros');
nueva_imagen = BW2;
nueva_imagen (~ máscara) = 0;
B = bwbo limits (nueva_imagen); % solo puede aceptar 2 dimensiones imshow (new_image, 'parent', app. UIAxes2);
mantener (app. UIAxes2, 'on');
límites fronterizos (B);
set (gca, 'YDir', 'reverse');
pulmonArea = bwarea (nueva_imagen);
app. PixelAreaEditField. Value = lungArea;
fin
fin
función FilterThresholdSliderValueChanged (aplicación, evento)
app.threshold = app. FilterThresholdSlider. Value;
fin
function AreastoExtractDropDownValueChanged (aplicación, evento) stringNumber = app. AreastoExtractDropDown. Value;
app.regionsToExtract = str2double (stringNumber);
fin
fin
Recomendado:
MATLAB Easy Face Detection: 4 pasos

MATLAB Easy Face Detection: el objetivo principal de este instructivo es mostrar lo fácil que será el procesamiento de imágenes. Con la ayuda de MATLAB, la detección y el seguimiento de rostros ha sido un campo de investigación importante y activo, por eso voy a explicar cómo se puede hacer con
Cómo codificar y publicar Matlab 2016b en Word (guía para principiantes): 4 pasos

Cómo codificar y publicar Matlab 2016b en Word (Guía para principiantes): Matlab es un programa de lenguaje de alto rendimiento que se utiliza para calcular resultados técnicos. Tiene la capacidad de integrar imágenes, cálculos y programación de una manera fácil de usar. Con este programa, el usuario puede publicar problemas y soluciones
Controlar Raspberry Pi con Matlab: 5 pasos

Controlando Raspberry Pi usando Matlab: Oye, este tutorial trata sobre cómo controlar tu Raspberry Pi sin cabeza usando matlab. Es posible que deba instalar matlab a la última versión para admitir la placa raspberry pi más nueva
Controlador robótico ROS basado en Matlab: 9 pasos
Controlador robótico ROS basado en Matlab: desde que era un niño, siempre he soñado con ser Iron Man y todavía lo hago. Iron Man es uno de esos personajes que es realistamente posible y, en pocas palabras, aspiro a convertirme en Iron Man algún día, incluso si la gente se ríe de mí o dice que es imposible
Mejoras en la programación de trenes en MATLAB: 6 pasos

Mejoras en la programación de trenes en MATLAB: Objetivo: El objetivo de este sistema programado es observar un Arduino a pequeña escala y aplicar la codificación a una escala mayor para mejorar potencialmente ciertas características de seguridad de los sistemas de Amtrak Railroad. Para hacer esto, hemos agregado un sensor de humedad del suelo
