Tabla de contenido:
- Paso 1: iniciar la interfaz gráfica de usuario (GUI)
- Paso 2: carga y lectura de imágenes de resonancia magnética en MATLAB
- Paso 3: filtrado de imágenes
- Paso 4: Aislamiento del tumor a través de una máscara elíptica
- Paso 5: descripción del tumor
- Paso 6: análisis de las propiedades físicas de los tumores
- Autor John Day day@howwhatproduce.com.
- Public 2024-01-30 08:42.
- Última modificación 2025-01-23 14:39.
Por: Madhumita Kannan, Henry Nguyen, Ashley Urrutia Avila, Mei Jin
Este código MATLAB es un programa para detectar el tamaño, la forma y la ubicación exactos de un tumor que se encuentra en las imágenes por resonancia magnética del cerebro de un paciente. Este programa está diseñado para trabajar originalmente con la detección de tumores en escáneres de resonancia magnética del cerebro, pero también se puede utilizar para el diagnóstico de cáncer en otros escáneres de órganos.
Las siguientes instrucciones describirán primero los métodos para el análisis de imágenes mediante el filtrado y la limpieza de la resonancia magnética, mediante la binarización, el filtrado medio y las ventanas deslizantes. A continuación, le indicará cómo aislar el tumor con una máscara elíptica pregenerada y filtrarlo más para delinear el perímetro de la forma del tumor.
Una vez que se detecta el tumor, las instrucciones describen con más detalle cómo incorporar este programa en una interfaz gráfica de usuario (GUI). A lo largo de estas instrucciones, se adjuntarán el código y los archivos correspondientes para ayudar a explicar cómo funciona este análisis de resonancia magnética.
Algunas cosas que debe conocer, descargar y tener listas antes de continuar con este instructivo: 1. Asegúrese de tener descargada la última versión de MATLAB. Puede instalar R2018b aquí:
2. Para ejecutar este programa, debe tener acceso a los archivos de exploración cerebral de MRI. Aunque siempre se pueden encontrar algunas en las imágenes de Google, se pueden realizar análisis exhaustivos y precisos a partir de imágenes adecuadas de varias capas de escáneres cerebrales para cada paciente. Puede acceder a los archivos de 20 pacientes diferentes con glioblastoma antes y después del tratamiento desde esta base de datos:
3. El enfoque de este programa y los diversos métodos que guían este proyecto se describen en este documento de investigación:
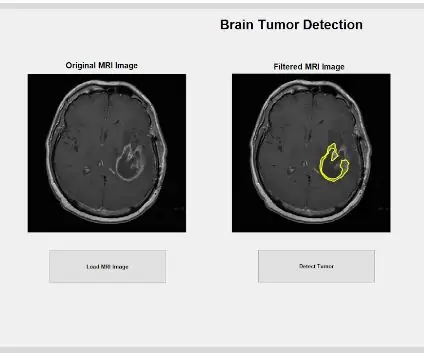
Paso 1: iniciar la interfaz gráfica de usuario (GUI)
El primer paso sería crear e iniciar la interfaz gráfica de usuario, GUI. Esto se puede hacer escribiendo guía en la ventana de comandos, presionando Intro y creando una nueva GUI. Una vez que se completa este paso, puede comenzar a crear funciones como ejes, texto estático, editar texto y botones de comando que se mostrarán una vez que se ejecute el programa y el usuario pueda interactuar. Estas funciones se pueden editar y manipular a través del inspector de propiedades, sin embargo, la característica más importante que debe modificarse al crear estas funciones es el nombre de la etiqueta. Es importante cambiar el nombre de la etiqueta de cada función que se implemente porque nos permitirá crear una función de devolución de llamada distinguible. Una vez que esté satisfecho con el diseño de su GUI, puede pasar a cargar los archivos DICOM que se mostrarán dentro de la GUI.
Paso 2: carga y lectura de imágenes de resonancia magnética en MATLAB
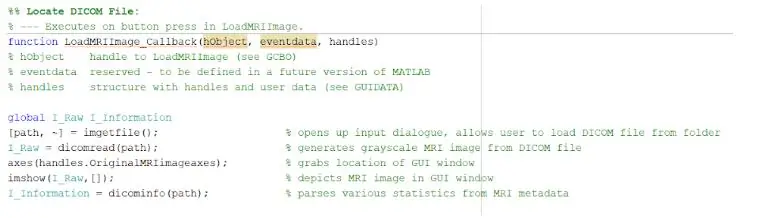
Para cargar los archivos DICOM, tendría que inicializar correctamente la función de devolución de llamada que se ejecutaría al presionar el botón "Cargar imagen de resonancia magnética". Una vez que esto se haya completado, debe crear una variable global que muestre la imagen en los ejes de las manijas donde desea que se muestre la imagen de resonancia magnética original. Las imágenes de resonancia magnética descargadas de la base de datos son todos archivos con formato DICOM que deben cargarse en su directorio MATLAB. Busque el archivo usando imgetfile para cargarlo en el programa. Las imágenes se leen usando la función incorporada de MATLAB "dicomread", y la primera imagen sin procesar de cada archivo se incrusta en los ejes de la GUI izquierda usando imshow.
La función incorporada de MATLAB 'dicominfo' también es extremadamente útil para abordar toda la información de cada archivo dicom de MRI. Utilizamos esta función para extraer toda la información descriptiva de los pacientes, como su sexo, edad, peso y altura. Esta función también le proporciona el orden de la pila que es útil para la implementación del programa dentro de la interfaz gráfica de usuario. Creamos variables para cada una de la información descriptiva de los pacientes que se utilizarán para la GUI cuando se presione el botón de detección.
Paso 3: filtrado de imágenes

Una vez que se ha cargado y leído el archivo DICOM de la imagen sin procesar, la imagen debe convertirse de escala de grises a una forma binarizada que consta solo de píxeles en blanco y negro Usamos la función 'imbinarizar' para crear una imagen binaria a partir de la imagen sin procesar mediante controlar aspectos del umbral adaptativo a un valor de sensibilidad de 0,59. El factor de sensibilidad de umbral predeterminado, 0.5, era bajo y no podía detectar las manchas y manchas más brillantes de la imagen, por lo que lo aumentamos a 0.59.
Luego, la imagen binarizada se procesa a través de un filtro de mediana utilizando la función "medfilt2" porque la imagen binarizada es bidimensional. Configuramos cada píxel de salida para que contenga el valor mediano en la vecindad de 5 x 5 alrededor del píxel correspondiente en la imagen binarizada de entrada. Esto reduce el ruido y conserva los bordes en un cuadrado de 5 x 5 alrededor de cada píxel. A continuación, aplicamos una ventana deslizante usando "strel", para crear un elemento estructurante plano en forma de disco con un radio de vecindad de 2 para identificar cada píxel de origen central, en cada vecindario de disco. Utilizamos un elemento de estructura de disco porque estamos analizando cada punto circular y los píxeles dentro de cada punto, por lo que un elemento de forma de disco es más útil.
Una vez que se ha filtrado la imagen, se puede limpiar con la función "imclose" para eliminar los puntos negros entre los píxeles blancos filtrados en la imagen y cerrar todos los espacios a su alrededor. La imagen completamente procesada se puede trazar en la segunda subtrama de la figura preasignada, lo que permite una comparación entre la imagen sin procesar y la imagen filtrada.
Paso 4: Aislamiento del tumor a través de una máscara elíptica


Los puntos brillantes del tumor se pueden aislar de la imagen filtrada principal a través de una máscara elíptica pregenerada. Para crear esta máscara, debe conocer el tamaño de la imagen de resonancia magnética sin procesar original y, utilizando su longitud de fila y columna, como coordenadas xey, respectivamente, asignar las coordenadas centrales para la elíptica. Establecemos el eje y como un eje mayor con un radio de 50 unidades desde el centro y el eje menor con un radio de 40 unidades desde el centro.
Usamos la función 'meshgrid' de MATLAB para generar un plano cartesiano con coordenadas de cuadrícula bidimensionales basadas en las coordenadas contenidas en los vectores desde 1 hasta la longitud del eje x, y desde 1 hasta la longitud del eje y de la imagen.. Col es una matriz donde cada fila es una copia del eje x, y Row es una matriz donde cada columna es una copia del eje y. La cuadrícula cartesiana representada por las coordenadas Col y Row tiene filas de longitud (1: Y_Size) y columnas de longitud (1: X_Size). Utilice los índices de Col y Row generados por la cuadrícula cartesiana para determinar la ecuación de la elipse según el radio predeterminado y las coordenadas centrales. El contorno elíptico ahora se puede rellenar con los píxeles blancos que se encuentran en las manchas tumorales.
Utilizando la máscara elíptica pregenerada, podemos recortar el tumor específico que desea analizar de la imagen filtrada. La máscara elíptica detecta qué puntos encajan lógicamente dentro del contorno de la elipse y lo acepta como un punto en la imagen filtrada para ser aceptable como tumor. La función "bwareafilt" luego filtra todos los demás objetos fuera de este tumor detectado de la imagen. Usamos una ventana específica de 500 por 4000 empíricamente basada en las dimensiones de todas las imágenes. Luego aplicamos otra ventana deslizante con "strel" como un elemento estructurante en forma de disco plano de un radio de vecindad mayor de 6, para cerrar los espacios entre cada píxel blanco central dentro del tumor detectado. La mancha del tumor detectada se limpia aún más usando "imclose" para eliminar aún más los píxeles negros y rellenar todos los agujeros con "imfill". Este tumor procesado se puede mostrar en la tercera subparcela del gráfico preasignado para proporcionar una comparación entre el tumor aislado y las imágenes originales y filtradas de la resonancia magnética.
Paso 5: descripción del tumor
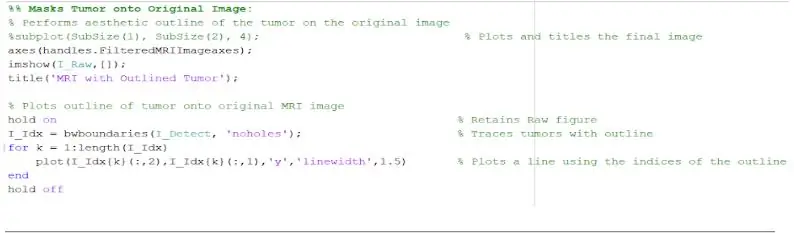
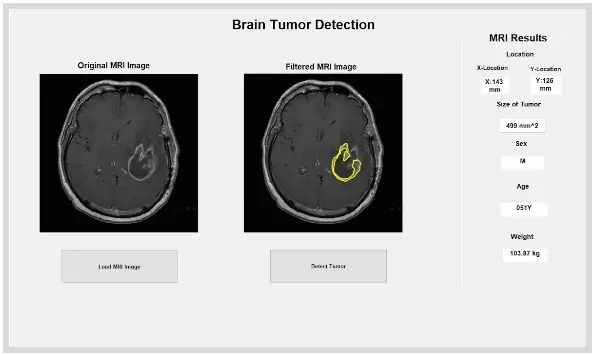
Ahora que el tumor está aislado con la máscara, se puede delinear y mostrar en la imagen original para mostrar su ubicación exacta. Para hacer esto, usamos la función "bwbo limits" para rastrear el tumor detectado previamente con un contorno. Especificamos el contorno para no incluir los orificios dentro del objeto del tumor a medida que se perfila. Esto se puede trazar en la imagen sin procesar original, utilizando un bucle "para" que traza el contorno alrededor del tumor utilizando los índices de la línea con un ancho de línea de 1,5 píxeles. Luego, este contorno se traza en la imagen sin procesar, que muestra el tamaño exacto y la ubicación del tumor, en relación con la resonancia magnética original.
Paso 6: análisis de las propiedades físicas de los tumores
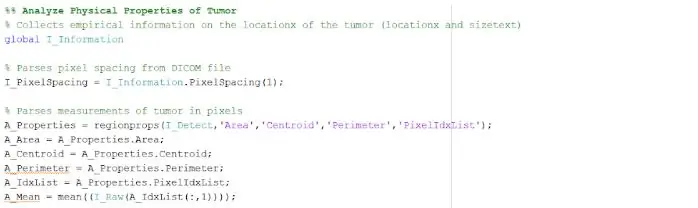
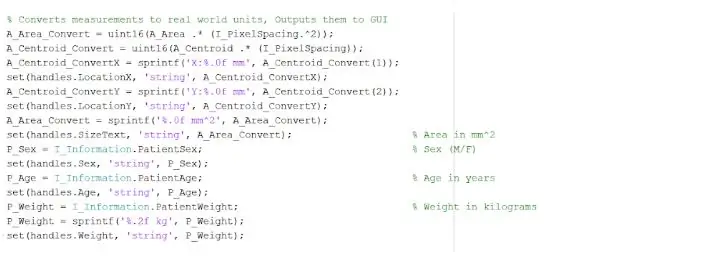
El lugar aislado y delineado puede proporcionarnos información útil sobre el tamaño, el área y la ubicación del tumor. Usamos la función "regionprops" para detectar las propiedades del tumor que pertenecen al área, perímetro, centroides y el valor del índice de píxeles. Este valor de índice de píxeles nos da las unidades del mundo real para cada píxel de cada imagen, únicas para cada escaneo. Estas propiedades luego se pueden convertir en unidades de milímetros del mundo real. La información empírica que nos proporciona el programa es única para cada resonancia magnética y es extremadamente útil para determinar el tamaño, la ubicación y el tipo de tumor, que los usuarios pueden analizar e incorporar en la interfaz gráfica de usuario.
Recomendado:
Sombrero parlante con detección de sacudidas con Circuit Playground Express: 12 pasos (con imágenes)

Shake Detecting Talking Hat con Circuit Playground Express: ¡Este sencillo y rápido tutorial te enseña cómo hacer un sombrero parlante! Respondería con una respuesta cuidadosamente procesada cuando "hicieras" una pregunta, y tal vez podría ayudarte a decidir si tienes alguna preocupación o problema. En mi clase de Wearable Tech, yo
Una caja de resonancia de bolsillo: 6 pasos

Una caja de resonancia de bolsillo: este dispositivo no solo cabe en un bolsillo sino que también produce varios tonos musicales similares a los de una gaita (en mi opinión) mediante varias combinaciones de seis pulsadores. Obviamente, es solo un artilugio para divertir a los niños; sin embargo, es principio
Caja de resonancia Raspberry Pi: 3 pasos

Caja de resonancia Raspberry Pi: El propósito de este proyecto es crear una caja de resonancia con una Raspberry Pi, para cualquier sonido. Yo personalmente lo creé con el propósito de crear un ambiente de caja de resonancia de Dungeons and Dragons. Sin embargo, puede usar esto para cualquier cosa si lo personaliza SETUPFor this p
Prototipo de caja de resonancia Arduino-Raspberry Pi: 9 pasos (con imágenes)

Prototipo de caja de resonancia Arduino-Raspberry Pi: la caja de resonancia prototipo creada con un Arduino y una Raspberry Pi está destinada a ser una forma sencilla de reproducir 4 sonidos o ruidos diferentes mientras se tiene la opción de cambiar conjuntos de sonidos con una perilla y mostrar el conjunto de sonidos actual con un Pantalla LCD. * Por favor
Guardián V1.0 --- Actualización de la cámara de mirilla de la puerta con Arduino (funciones de grabación por detección de movimiento y descarga eléctrica): 5 pasos

Guardián V1.0 ||| Actualización de la cámara de mirilla de puerta con Arduino (funciones de grabación por detección de movimiento y descarga eléctrica): he pedido una cámara de mirilla, pero cuando la uso, me di cuenta de que no hay una función de grabación automática (activada por detección de movimiento). Luego comencé a investigar cómo funciona. Para grabar un video, debes 1- mantener presionado el botón de encendido casi 2 segundos
